Las informaciones obtenidas de los estudios de electroforesis se han estructurado en tres tablas: la tabla ELECSTUDIES, que contiene, para cada especie, informaciones generales acerca de los estudios llevados a cabo en diferentes poblaciones de esa especie; la tabla ELECDAT, que contiene los datos por locus y por estudio; y la tabla ELECSUB, que contiene los alelos que han sido detectados para cada locus.
En conjunto, estas tablas proporcionan información acerca de la estructura genética y de la variabilidad de las poblaciones de peces, tanto naturales como cultivadas. Estas informaciones son importantes para la selección de especies o de estirpes para la acuicultura. Asimismo, facilitarán los programas de gestion y de conservación de los stocks naturales.
A medida que se iban incorporando nuevos datos a esta tabla, ha sido posible evidenciar las actuales lagunas, tanto en los conocimientos (es decir, las especies importantes que todavía están poco estudiadas) como en los métodos y los formatos para reportar a FishBase, de la manera más adecuada, la caracterización genética de las distintas especies.
Las tablas contienen las frecuencias alélicas de los estudios electroforéticos de poblaciones tanto salvajes como cultivadas. También contienen informaciones acerca de los enzimas, del número total de loci estudiados, de los tejidos y los sistemas tampón empleados, de los valores de heterocigosis y de las proporciones de loci polimórficos.
Locality and Country [Localidad y País]: Indica el lugar de captura de los especímenes.
Total loci [Número total de loci]: Indica el número total de loci examinados.
Observed heterozygosity [Heterocigosis observada]: Indica, para un número dado de loci, la proporción de individuos heterocigotos en una población,. Se dice que un individuo es homocigoto si posee dos alelos idénticos en un mismo locus, e inversamente se dice que es heterocigoto si son dos alelos diferentes.
Expected heterozygosity [Heterocigosis esperada]: Indica la proporción de individuos heterocigotos, calculada a partir de las frecuencias alélicas conocidas, suponiendo que la población está en equilibrio de Hardy-Weinberg. Se calculan por locus, población y especie, y permiten apreciar el potencial para una cría selectiva (véase Fig. 53).
Polymorphic loci [Loci polimórficos]: Indica el número de loci polimórficos presentes en una muestra, en relación con el número total de loci examinados (véase Fig. 54). Para estandardizar los datos, se ha aplicado el criterio del 95%, según el cual un locus se considera polimórfico si la frecuencia alélica más fuerte no sobrepasa el valor 0.95. En los casos en los que se aplica el criterio del 99%, constará una indicación en Comment [Comentario].
Enzyme [Enzima]: Indica los nombres, las abreviaturas y los códigos numéricos estandarizados para los enzimas y otras proteínas habitualmente analizadas en los trabajos de genética de peces. Los nombres y los códigos utilizados corresponden a la nomenclatura recomendada por el comité de la International Union of Biochemistry (Shaklee et al. 1990).
Locus (plural: Loci): Indica la posición específica o la localización de un gen sobre el cromosoma. Un gen es una porción específica de ADN (ácido desoxirribonucleico) que ocupa un locus. Un locus es monomorfo si sólo se conoce un único alelo del gen situado en ese locus, y es polimórfico cuando se conocen varios alelos. Si dos loci o más están implicados en la producción de distintas formas de una misma proteína (isozimas), al locus correspondiente a la proteína con más carga positiva se le atribuye el número 1, 2 al siguiente, etc. A veces, el locus se designa con letras, designándose con una A al locus que codifica para la proteína con más carga positiva, B al siguiente y así sucesivamente.
Tissue [Tejido]: El tipo de tejido elegido para la electroforesis puede serlo entre los siguientes tipos: skeletal muscle [músculo esquelético]; visceral muscle [músculo visceral]; heart [corazón]; kidney [riñón]; liver [hígado]; blood [sangre]; mucus [mucus]; eye lens [cristalino]; whole body [totalidad del cuerpo]; others [otros]. La elección del tipo de tejido se precisa en Comment [Comentario].
Method used [Método empleado]: Indica el tipo de método electroforético utilizado, de entre las siguientes alternativas: starch gel [gel de almidón]; polyacrylamide gel [gel de poliacrilamida]; sodium dodecyl sulfate [dodecil sulfato sódico]; other methods [otros métodos]. La electroforesis en gel es uno de los métodos más comunes para estudiar la variación genética de los individuos, tanto a nivel de la estirpe como de la especie.
Buffer system [Sistema tampón]: Indica qué tampón ha sido empleado durante la electroforesis para obtener una separación clara de las proteínas y de los enzimas específicos. Los 15 tampones habitualmente más utilizados están descritos por Boyer et al. (1963), Ridgway et al. (1970), Shaw y Prasad (1970), Selander et al. (1971), y Clayton y Tretiak (1972).
pH: Indica el pH del tampón utilizado.
Samples [Muestras]: Indica el número de especímenes estudiados por localidad o por población.
Allele [Alelo]: Indica uno de los alelos (es decir, una de las formas alternativas) del gen estudiado. Los alelos se identifican durante la electroforesis por sus productos proteicos (enzimas). En un zymograma, la mobilidad electroforética relativa de los enzimas se expresa numéricamente. Las mobilidades relativas se calculan asignando el valor 100 al alelo anódico más común (o -100 para un locus catódico). El signo negativo se atribuye a los alelos con mobilidad catódica.
Allele frequency [Frecuencia alélica],: Se calcula para un locus determinado mediante la siguente fórmula: frecuencia alélica A=2. (frecuencia genotípica AA) + (frecuencia genotípica Aa) / 2n, donde n = número de individuos estudiados.
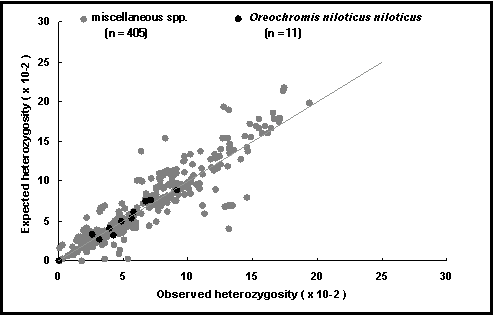
Fig. 53. Predicciones de heterocigosis en función de la heterocigosis observada en Oreochromis niloticus niloticus (puntos negros) y diversas especies de peces (puntos blancos). La pendiente de la diagonal es 1, e identifica los puntos en los cuales la heterocigosis esperada es igual a la observada. Los valores muy por encima de ella pueden deberse a la consanguinidad, y los valores muy por debajo al cruce de estirpes.
Las tablas ELECSTUDIES, ELECDAT y ELECSUB contienen actualmente más de 11.000 registros (cada registro trata los alelos de un solo locus) de frecuencias alélicas de más de 900 estudios y más de 800 poblaciones/estirpes de peces. La actualización de estas tablas es fruto de la colaboración de D.O.F. Skibinski y de su equipo, a partir de referencias que ellos han repertoriado (Skibinski et al. 1991), y constituye la mayor recopilación de la variabilidad genética de los peces.
A partir de esta tabla pueden construirse varios gráficos:
La relación entre la heterocigosis esperada y la observada (véase Fig. 53), que analiza si la variabilidad genética (H y P) se ha reducido en las poblaciones en cautividad comparadas con las poblaciones salvajes;
La relación entre el contenido de ADN celular y la ordenación filogenética de Fishes of the world de Nelson (1994) (véase la Fig. 51);
La relación entre el número de cromosomas y el contenido en ADN celular; y
La relación entre el contenido de ADN celular y el índice de forma de la aleta caudal (para una explicación de este gráfico véase la Fig. 52 y el Recuadro 29).
Todos estos gráficos pueden ser mostrados a través de la vista GENETICS, y se refieren a la especie seleccionada. O bien, pueden ser mostrados desde la ventana REPORTS y después GRAPHS.
Las principales referencias bibliográficas ya citadas son: Winans (1980), McAndrew y Majumdar (1983), Macaranas et al. (1986, 1995), van der Bank et al. (1989), Carvalho et al. (1991) y Pouyaud y Agnèse (1995).
Compilar la totalidad de las frecuencias alélicas y las informaciones relacionadas ya publicadas es un reto difícil, que implica la resolución de problemas como la falta de estandarización entre las publicaciones, que impide el reagrupamiento de los datos (Agustin et al. 1993, 1994).
Hacer clic en el botón Biology de la vista SPECIES, y después en el botón Genetics de la vista BIOLOGY, y sobre el botón Allele frequencies de la ventana GENETICS. Hacer doble clic sobre una de las líneas de la vista LIST OF AVAILABLE ELECTROPHORETIC STUDIES permite mostrar las informaciones relativas al estudio señalado.
Haciendo clic sobre el botón Electrophoretic data de la vista INFORMATION ON HETEROZYGOTY AND POLYMORPHISM se muestra la lista de loci (enzimas). Un doble clic sobre una de las líneas permite mostrar las informaciones del locus seleccionado.
Un clic sobre el botón Allele Frequencies de la vista LOCUS INFORMATION muestra las frecuencias alélicas (‹ La tabla ELECDAT ›).
Nuestro agradecimiento para R.E. Brummett, A.E. Eknath, G.C. Mair, J.G. McGlade, D. Pauly, R.S.V. Pullin y D.O. Skibinski, por sus consejos acerca de la estructura y el contenido de esta tabla.
Agustin, L.Q., R. Froese, A.E. Eknath and R.S.V. Pullin. 1993. Documentation of genetic resources for aquaculture - the role of FishBase, p. 63-68. In D. Penman, N. Roongratri and B. McAndrew (eds.) International Workshop on Genetics in Aquaculture and Fisheries Management. ASEAN-EEC Aquaculture Development and Coordination Programme, Bangkok, Thailand.
Agustin, L.Q., M.L.D. Palomares and G.C. Mair. 1994. FishBase: a repository of genetic information on fish. Poster presented at the Fifth International Symposium on Genetics in Aquaculture, 19-25 June 1994, Dalhousie University, Halifax, Nova Scotia, Canada.
Boyer, S.H., D.C. Fainer and E.J. Watson-Williams. 1963. Lactate dehydrogenase variant from human blood: evidence for molecular subunits. Science 141:642-643.
Carvalho, G.R., P.W. Shaw, A.E. Magurran and B.H. Seghers. 1991. Marked genetic divergence revealed by allozymes among populations of the guppy Poecilia reticulata (Poeciliidae), in Trinidad. Biol. J. Linn. Soc. 42:389-405.
Clayton, J.W. and D.N. Tretiak. 1972. Amine-citrate buffers for pH control in starch gel electrophoresis. J. Fish. Res. Board Can. 29:1169-1172.
Macaranas, J.M., N. Taniguchi, M.J.R. Pante, J.B. Capili and R.S.V. Pullin. 1986. Electrophoretic evidence for extensive hybrid gene introgression into commercial Oreochromis niloticus (L.) stocks in the Philippines. Aquacult. Fish. Manage. 17:249-258.
Macaranas, J.M., L.Q. Agustin, M.C.A. Ablan, M.J.R. Pante, A.E. Eknath and R.S.V. Pullin. 1995. Genetic improvement of farmed tilapias: biochemical characterization of strain differences in Oreochromis niloticus. Aquaculture International 3:43-54.
McAndrew, B.J. and K.C. Majumdar. 1983. Tilapia stock identification using electrophoretic markers. Aquaculture 30:249-261.
Nelson, J.S. 1994. Fishes of the world. 3rd edition. John Wiley and Sons, New York. 600 p.
Pouyaud, L. and J.-F. Agnèse. 1995. Phylogenetic relationships between 21 species of three tilapiine genera Tilapia, Sarotherodon and Oreochromis using allozyme data. J. Fish Biol. 47(1):26-38.
Ridgway, G.J., S.W. Sherburne and R.D. Lewis. 1970. Polymorphism in the esterases of Atlantic herring. Trans. Am. Fish. Soc. 99:147-151.
Selander, R.K., M.H. Smith, S.Y. Yang, W.E. Johnson and J.B. Gentry. 1971. Biochemical polymorphism and systematics in the genus Peromyscus. I. Variation in the old field mouse (Peromyscus polionotus). Studies in Genetics VI. Univ. Texas Publ. 7103:49-90.
Shaklee, J.B., F.W. Allendorf, D.C. Morizot and G.S. Whitt. 1990. Gene nomenclature for protein-coding loci in fish. Trans. Am. Fish. Soc. 119:2-15.
Shaw, C.R. and R. Prasad. 1970. Starch gel electrophoresis of enzymes - a compilation of recipes. Biochem. Genet. 4:297-320.
Skibinski, D.O.F., M. Woodwark and R.D. Ward. 1991. The protein diversity database. University College of Swansea, Singleton Park, Swansea, Wales and CSIRO Division of Fisheries, Tasmania, Australia. 16 p.
van der Bank, F.H., W.S. Grant and J.T. Ferreira. 1989. Electrophoretically detectable genetic data for fifteen southern African cichlids. J. Fish Biol. 34:465-483.
Winans, G.A. 1980. Geographic variation in the milkfish Chanos chanos. I. Biochemical evidence. Evolution 34(3):558-574.
Christine Casal y Liza Agustin